[Statistics] ANOVA

ANOVA(분산분석)
- 두 개 이상의 집단에 대한 평균 차이를 집단 분산에 비교하여 분석하는 기법은 anova이다.
- 각 표본의 수는 같지 않아도 된다.
- H0: 모든 집단의 평균이 동일하다. vs H1: 적어도 한 집단의 평균이 다른 집단들과 다르다.
- F 통계량을 통해 검정한다.
- 설명변수(요인; factor)는 범주형(categorical)이어야 하고, 종속변수는 연속형(continuous)이어야 한다.
- 요인의 수준: level. 각 집단을 의미함.
- ANOVA는 요인의 수와 종속변수 수에 따라 구분할 수 있다.
- one-way ANOVA(일원분산분석): 독립변수(요인) 1개, 종속변수 1개
- two-way ANOVA(이원분산분석): 독립변수(요인) 2개, 종속변수 1개
- 이 경우 두 독립변수 간 교호작용에 대한 검증이 반드시 진행되어야 한다.
- 교호작용이란, 두 변수 간 상관관계가 있는지 확인하는 것이며, 교호작용이 없을 경우 주효과 검정을 진행한다.
- n-way ANOVA: 독립변수(요인) 3개 이상, 종속변수 1개
- MANOVA(다변량분산분석): 독립변수 1개 이상, 종속변수 2개 이상
| n-way ANOVA | Model | Description |
|---|---|---|
| one-way ANOVA | y ~ x1 | y is explained by x1 only |
| two-way ANOVA | y ~ x1 + x2 | y is explained by x1 and x2 |
| two-way ANOVA | y ~ x1 * x2 | y is explained by x1, x2 and interaction between them |
| three-way ANOVA | y ~ x1 + x2 + x3 | y is explained by x1, x2 and x3 |
| MANOVA | y1 + y2 ~ x1 | y1, y2 are explained by x1 |
- ANOVA 분석을 진행한 후 사후 분석(Post Hoc)을 추가로 진행할 수 있다.
- 적어도 한 집단에서 평균 차이가 있다면, 어떤 집단들에 대해 평균 차이가 존재하는지 알아보기 위해 실시한다. (유의미한 차이가 없을 경우 사후분석을 진행할 필요 없다.)
- 사전 가설 설정없이 ANOVA를 시행한 경우, 탐색적으로 평균 차이가 나는 집단을 살펴보기 위해 시행하는 방법이다.
- 조합 가능한 모든 쌍에 대해 비교를 하므로 과잉검증으로 인해 FWER(Family-wise Error Rate)가 증가할 수 있다.
- FWER: 여러 개의 가설 검정을 할 때 적어도 하나의 가설에서 1조 오류가 발생할 가능성을 의미
- 가설검정을 n번 했을 때 n번 모두 1종 오류가 발생하지 않을 확률(FWER)은 $1-0.95^n$
- 본페로니 교정(Bonferroni correction), 튜기(Tukey)의 HSD 방법, 피셔의 최소유의차(LSD) 등을 사용
- MANOVA 분석 이후, 일변량 분석을 실시하여 각 범주 내에서 어떤 변수가 유의미한 차이를 가져왔는지 확인할 수 있다.
- ANOVA를 수행하기 전, 만족해야 하는 가정은 다음과 같다.
- 독립성: 자료의 추출은 독립적으로 이루어졌다.
- 등분산성: 모든 집단의 모분산은 동일하다.
- 정규성: 자료의 모집단 분포는 정규분포를 따른다. — 집단별로 실시하여 확인
| distribution before transformation | transformation function |
|---|---|
| left | x^3 |
| mild left | x^2 |
| mild right | sqrt(t) |
| right | ln(x) |
| severe right | 1/x |
Excercise
import pandas as pd
import warnings
warnings.filterwarnings(action='ignore')
import scipy
data = pd.read_csv('/patent_18.csv')
data.info() # 사용할 컬럼만 추출하였습니다.
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 11448 entries, 0 to 11447
Data columns (total 32 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
4 사업화유무 11448 non-null object
9 pqi 11448 non-null float64
11 score 11448 non-null float64
14 예측분류 11448 non-null object
31 ipc 11448 non-null object
dtypes: float64(6), int64(13), object(13)
memory usage: 2.8+ MB
가정1: 독립성
data[['projectnumber','예측분류']].groupby('예측분류').agg(len) # 기타 칼럼은 생략함
| projectnumber | |
|---|---|
| 예측분류 | |
| BT(생명공학기술) | 3001 |
| CT(문화기술) | 274 |
| ET(환경기술) | 3123 |
| IT(정보기술) | 3377 |
| NT(나노기술) | 1415 |
| ST(우주항공기술) | 258 |
- random sampling을 진행한 경우 독립성을 만족한 것으로 본다.
- 따라서 집단별 최대 300개의 데이터를 갖도록 랜덤 추출을 진행하였다.
def gpSampling(data, group, group_N, random_num=123) :
import pandas as pd
df_data_sp = data.sample(frac=1,random_state=random_num,replace = False).groupby(by = group).head(group_N) # group에 따라 일정 개수의 샘플을 복원 추출하여 사용한다.
return df_data_sp
data_sampled = gpSampling(data, '예측분류', 300)
가정2: 정규성
- 그래프: 가장 빠르고 쉽게, 직관적으로 확인할 수 있는 방법이다. 집단별로 정규성을 만족하는지 확인한다.
# 1. histogram: 한 쪽으로 치우치면 정규분포라 하기 어려움
# 2. kernel density: 한 쪽으로 치우치면 정규분포라 하기 어려움
# 3. q-q plot: 대각선을 기준으로, 점들이 선형을 이루며 붙어 있으면 정규성을 이룬다고 판단.
# 우선 히스토그램으로 정규성을 확인하였다.
f,axes = plt.subplots(2,3, figsize=(20,10))
sns.distplot(data_sampled['pqi'][data_sampled['예측분류']=='CT(문화기술)'], ax=axes[0,0])
sns.distplot(data_sampled['pqi'][data_sampled['예측분류']=='BT(생명공학기술)'], ax=axes[0,1])
sns.distplot(data_sampled['pqi'][data_sampled['예측분류']=='ET(환경기술)'], ax=axes[0,2])
sns.distplot(data_sampled['pqi'][data_sampled['예측분류']=='IT(정보기술)'], ax=axes[1,0])
sns.distplot(data_sampled['pqi'][data_sampled['예측분류']=='NT(나노기술)'], ax=axes[1,1])
sns.distplot(data_sampled['pqi'][data_sampled['예측분류']=='ST(우주항공기술)'], ax=axes[1,2])
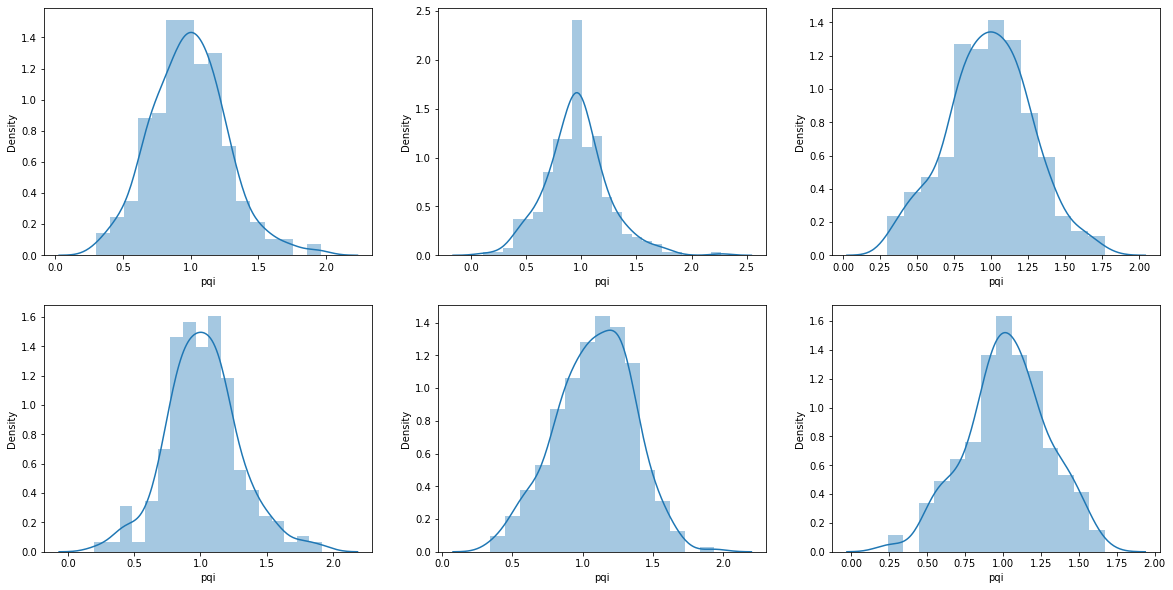
- 정규분포의 형태를 띈다고 할 수 있다.
f,axes = plt.subplots(2,3, figsize=(20,10))
sns.distplot(data_sampled['score'][data_sampled['예측분류']=='CT(문화기술)'], ax=axes[0,0])
sns.distplot(data_sampled['score'][data_sampled['예측분류']=='BT(생명공학기술)'], ax=axes[0,1])
sns.distplot(data_sampled['score'][data_sampled['예측분류']=='ET(환경기술)'], ax=axes[0,2])
sns.distplot(data_sampled['score'][data_sampled['예측분류']=='IT(정보기술)'], ax=axes[1,0])
sns.distplot(data_sampled['score'][data_sampled['예측분류']=='NT(나노기술)'], ax=axes[1,1])
sns.distplot(data_sampled['score'][data_sampled['예측분류']=='ST(우주항공기술)'], ax=axes[1,2])
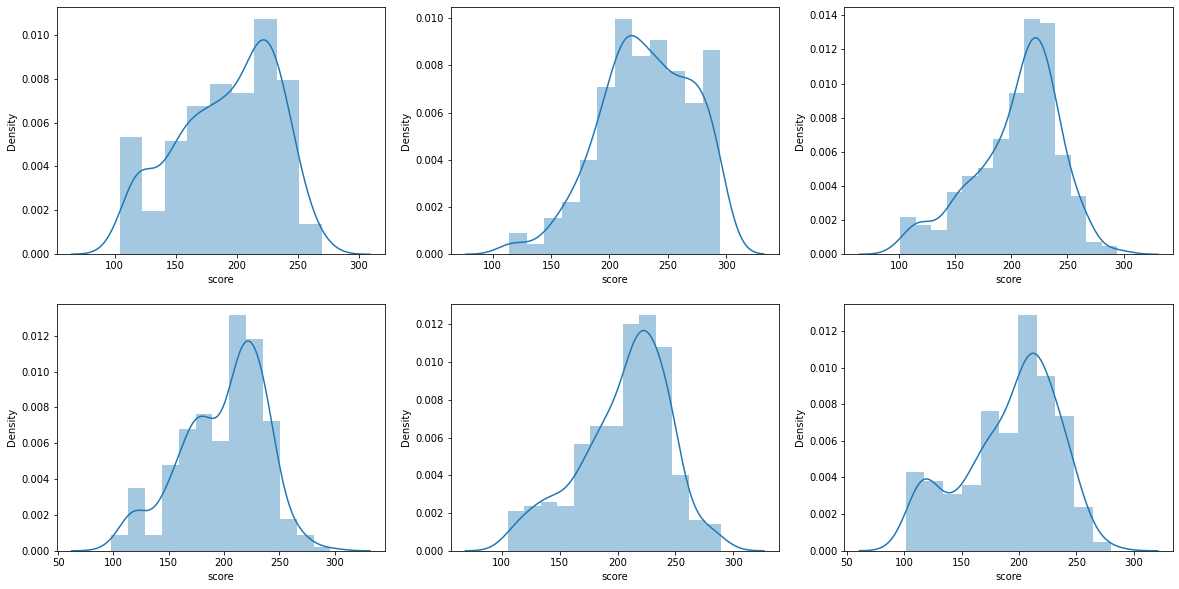
- 정규분포의 형태를 띈다고 할 수 있다.
- Shapiro-Wilk test: 오차항이 정규분포를 따르는지 알아보는 검정으로, 모든 독립변수에 대해 종속변수가 정규분포를 따르는지 알아보는 방법이다.
- 귀무가설을 기각하지 못한다는 것이 100% 정규성을 만족한다는 이야기는 아니므로, 참고하는 정도로만 본다.
- H0: 정규분포를 따른다. -> p-value가 0.05보다 클 경우 정규성을 가정
-
Kolmogorov-Smirnov test: EDF에 기반한 적합도 검정 방법으로, 자료의 평균/표준편차와 히스토그램을 표준정규분포와 비교한다.
# H0: 정규분포를 따른다. -> p-value가 0.05보다 클 경우 정규성을 가정 from scipy import stats print(stats.ks_2samp(x1, x2), stats.ks_2samp(x1, x3), stats.ks_2samp(x1, x4), sep="\n")Ks_2sampResult(statistic=0.0940632603406326, pvalue=0.1457968119870704) Ks_2sampResult(statistic=0.07072992700729927, pvalue=0.4433034063437392) Ks_2sampResult(statistic=0.1640632603406326, pvalue=0.0007587362449577029)
가정3: 등분산성
- Levene test: 유의수준 5% 하에서 p-value가 0.05보다 클 경우 귀무가설을 기각하지 못하여 등분산성을 만족한다고 할 수 있다.
scipy.stats.levene(
data_sampled.score[data_sampled.예측분류 == 'CT(문화기술)'],
data_sampled.score[data_sampled.예측분류 == 'BT(생명공학기술)'],
data_sampled.score[data_sampled.예측분류 == 'ET(환경기술)'],
data_sampled.score[data_sampled.예측분류 == 'IT(정보기술)'],
data_sampled.score[data_sampled.예측분류 == 'NT(나노기술)'],
data_sampled.score[data_sampled.예측분류 == 'ST(우주항공기술)'],)
LeveneResult(statistic=1.7932490047476697, pvalue=0.11104264617427342)
- Bartlett test: 유의수준 5% 하에서 p-value가 0.05보다 클 경우 귀무가설을 기각하지 못하여 등분산성을 만족한다고 할 수 있다.
scipy.stats.levene(
data_sampled.pqi[data_sampled.예측분류 == 'CT(문화기술)'],
data_sampled.pqi[data_sampled.예측분류 == 'BT(생명공학기술)'],
data_sampled.pqi[data_sampled.예측분류 == 'ET(환경기술)'],
data_sampled.pqi[data_sampled.예측분류 == 'IT(정보기술)'],
data_sampled.pqi[data_sampled.예측분류 == 'NT(나노기술)'],
data_sampled.pqi[data_sampled.예측분류 == 'ST(우주항공기술)'],)
LeveneResult(statistic=0.3786895075055586, pvalue=0.8636019543573566)
- 두 지표 모두 pvalue가 0.05보다 크므로 등분산성을 만족한다.
ANOVA
- 본 검정은 예측분류에 따른 pqi, score 값의 평균 차이를 검정하고자 하는 것이므로 MANOVA를 실시해야 하지만, 다른 검정을 위해서는 ANOVA를 수행할 수도 있다.
-
모든 집단별 표본수가 동일할 경우 균형설계자료
from statsmodels.formula.api import ols from statsmodels.stats.anova import anova_lm model = ols('pqi ~ 예측분류 + ipc', data_sampled).fit() # 수치형을 factor 형태로 표현하기 위해 C(변수) anova_lm(model) -
집단별 표본수가 동일하지 않을 경우 비균형설계자료
from statsmodels.formula.api import ols from statsmodels.stats.anova import anova_lm model = ols('pqi ~ 예측분류 + ipc', data_sampled).fit() anova_lm(model, typ=3) -
MANOVA
from statsmodels.multivariate.manova import MANOVA maov=MANOVA.from_formula('pqi + score ~ 예측분류', data=data_sampled) print(maov.mv_test())Multivariate linear model ================================================================ ---------------------------------------------------------------- Intercept Value Num DF Den DF F Value Pr > F ---------------------------------------------------------------- Wilks' lambda 0.2215 2.0000 1725.0000 3031.3782 0.0000 Pillai's trace 0.7785 2.0000 1725.0000 3031.3782 0.0000 Hotelling-Lawley trace 3.5146 2.0000 1725.0000 3031.3782 0.0000 Roy's greatest root 3.5146 2.0000 1725.0000 3031.3782 0.0000 ---------------------------------------------------------------- ---------------------------------------------------------------- 예측분류 Value Num DF Den DF F Value Pr > F ---------------------------------------------------------------- Wilks' lambda 0.8502 10.0000 3450.0000 29.1684 0.0000 Pillai's trace 0.1519 10.0000 3452.0000 28.3645 0.0000 Hotelling-Lawley trace 0.1739 10.0000 2584.7515 29.9791 0.0000 Roy's greatest root 0.1589 5.0000 1726.0000 54.8411 0.0000 ================================================================
Interpretation
Pr(>F)가 p-value로, 유의수준 5% 하에서 이 값이 0.05보다 작을 경우 통계적으로 유의미한 차이가 있다고 판단한다.- 위 결과를 보면 p-value가 0.0000으로, 예측분류에 따라 유의미한 차이가 있다는 것을 확인할 수 있다.
Post Hoc
from statsmodels.sandbox.stats.multicomp import MultiComparison
import scipy.stats
comp = MultiComparison(data_sampled.pqi, data_sampled.예측분류)
-
Bonferroni Correction
result = comp.allpairtest(scipy.stats.ttest_ind, method='bonf') result[0] -
Tuckey’s HSD
from statsmodels.stats.multicomp import pairwise_tukeyhsd hsd = pairwise_tukeyhsd(data_sampled['pqi'], data_sampled['예측분류'], alpha=0.05) hsd.summary() # p-value가 유의 수준보다 작을 경우 평균 차이가 유의미하다.
- ANOVA에 대해 더 자세히 알아보고자 하면 다음 내용을 참고한다.
- 가설
- H0: $mu_1=mu_2=…=mu_r$
- H1: $모든 mu_i는 같지 않다. i=1, 2, …, r$
-
기각역 혹은 p-value에 의한 검정
\[f_0>=F_\alpha(r-1,nT-r) \ 또는 \ p-value=P(F>=f_0)< \alpha 이면 \ H_0 기각 \\ f_0<F_\alpha(r-1,nT-r) \ 또는 \ p-value=P(F>=f_0)>= \alpha 이면 \ H_0 채택\] -
Two-way ANOVA Table
\[Y_{ijk}-\bar{Y_{...}}=(\bar{Y}_{i..}-\bar{Y}_{...})+(\bar{Y}_{.j.}-\bar{Y}_{...})+((\bar{Y}_{ij.}-\bar{Y}_{i..})-(\bar{Y}_{.j.}-\bar{Y}_{...}))+(Y_{ijk}-\bar{Y}_{ij.}) \\ Total = Factor A + FactorB+Interaction+Error\\SSTotal=SSA+SSB+SSAB+SSE\\~\\Y_{...}:전체 \ 평균\\Y_{i..}:요인A의 \ i번째 \ treatment의 \ 평균\\Y_{.j.}:요인B의 \ j번째 \ treatment의 \ 평균\\Y_{ij.}:요인A의 \ i번째 \ treatment \ 와 \ 요인B의 \ j번째 \ treatment의 \ 평균\]Source Sum of Squares Degrees of Freedom Mean Square F-statistic Factor A SSA a-1 MSA MSA/MSE Factor B SSB b-1 MSB MSB/MSE Interaction effect of A, B SSAB a-1 MSAB MSAB/MSE Error SSE ab(n-1) MSE Total SST nab-1
- 참고
- https://www.pythonfordatascience.org/factorial-anova-python/
- https://rfriend.tistory.com/tag/two-way ANOVA
- https://specialscene.tistory.com/19
- https://rfriend.tistory.com/tag/일원분산분석
- https://mindscale.kr/course/basic-stat-python/19/
- https://rfriend.tistory.com/118
- https://mindscale.kr/course/basic-stat-python/22/

댓글남기기