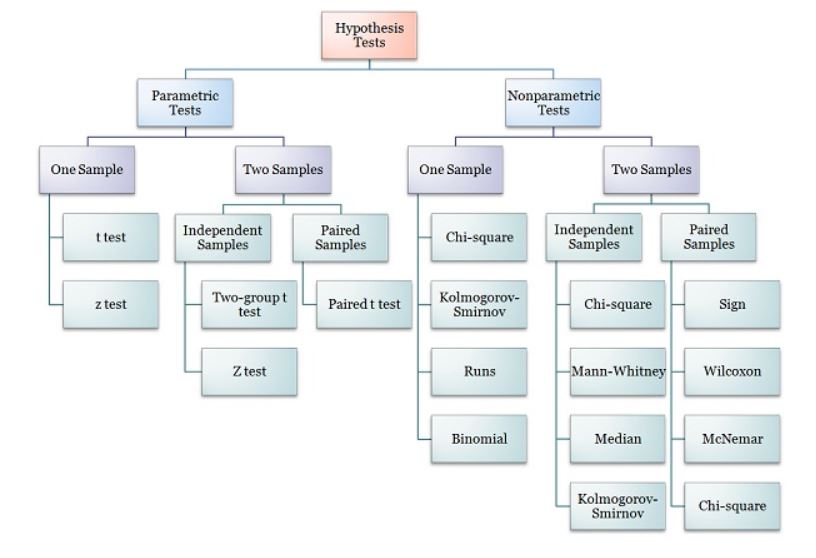
[Statistics] Non-parametric Test (비모수검정)
- 비모수 검정은 모수에 대한 가정을 전제로 하지 않고, 모집단의 형태와 관계없이 주어진 데이터에서 직접 확률을 계산하여 검정하는 방법이다.
- 일반적으로 rank(순위; 상대적 크기)를 이용하여 비교하며, 데이터 샘플 간 동일한 분포에서 나온 것인지를 검정할 수 있다.
- 비모수 검정을 실시할 때 귀무가설은, 데이터 샘플 간 동일한 분포를 가진 모집단에서 나왔으며, 따라서 동일한 population parameter을 가진다는 것이다.
- 따라서, $p-value \leq \alpha$인 경우 귀무가설을 기각하여, 다른 분포를 가진다는 것을 의미하고, 그 반대의 경우 동일 분포를 가진다는 것을 의미한다.
비모수 검정을 사용하는 경우
- 표본수가 적으면서 정규성을 만족하는 경우
- CLT(Central Limit Theorem)은 이론적으로 표본의 수가 많을 때 정규분포를 따른다는 정리로, 모수적 방법을 사용할 수 있게 된다.
- 변수가 명목척도이거나 서열척도인 경우
- 수치보다 상대적 크기가 의미있는 경우
모수 검정과 비교
| Basic For Comparison | Parametric Test | Nonparametric Test |
|---|---|---|
| Meaning | A statistical test, in which specific assumptions are made about the population parameter | A statistical test used in the case of non-metric independent variables |
| Basis of test statistic | Distribution | Arbitrary |
| Measurement level | Interval or ratio | Nominal or Ordinal |
| Measure of central tendency | Mean | Median |
| Information about population | Completely known | Unavailable |
| Applicability | Variables | Variables and Attributes |
| Correlation test | Pearson | Spearman |
비모수 검정 종류
- Mann-Whitney U test: 독립적인 샘플끼리 비교 (Student t-test의 비모수 버전)
- Wilcoxon signed-rank test: 짝지어진 샘플끼리 비교 (paired Student t-test의 비모수 버전)
- Kruskal-Wallis H and Friedman tests: 둘 이상의 샘플 비교 (ANOVA의 비모수 버전)
Exercise
- 가장 먼저, 데이터 셋을 생성하였다.
- 두 샘플은 서로 다른 분포를 가지고 있다. (아래의 경우 Gaussian 분포를 부여하였다.)
- 첫 번째 샘플은 mean 50, sd 5인 분포이며, 두 번째 샘플은 mean 51, sd 5인 분포이다.
# generate gaussian data samples from numpy.random import seed from numpy.random import randn from numpy import mean from numpy import std # seed the random number generator seed(1) # generate two sets of univariate observations data1 = 5 * randn(100) + 50 data2 = 5 * randn(100) + 51 # summarize print('data1: mean=%.3f stdv=%.3f' % (mean(data1), std(data1))) print('data2: mean=%.3f stdv=%.3f' % (mean(data2), std(data2))) -
Mann-Whitney U test
The two samples are combined and rank ordered together. The strategy is to determine if the values from the two samples are randomly mixed in the rank ordering or if they are clustered at opposite ends when combined. A random rank order would mean that the two samples are not different, while a cluster of one sample values would indicate a difference between them.
— Page 58, Nonparametric Statistics for Non-Statisticians: A Step-by-Step Approach, 2009.
- 검정을 위해 각 샘플 당 최소 20개 이상의 관측치가 필요하다.
from numpy.random import seed from numpy.random import randn from scipy.stats import mannwhitneyu # seed the random number generator seed(1) # generate two independent samples data1 = 5 * randn(100) + 50 data2 = 5 * randn(100) + 51 # compare samples stat, p = mannwhitneyu(data1, data2) print('Statistics=%.3f, p=%.3f' % (stat, p)) # interpret alpha = 0.05 if p > alpha: print('Same distribution (fail to reject H0)') else: print('Different distribution (reject H0)') - Wilcoxon Signed-Rank Test
- 두 샘플이 다른 어떠한 방식으로 인해 paired하게 측정된 데이터일 경우 사용한다.
- 예를 들어, 동일한 기계로부터 나온 두 개의 관측값이라든가, 동일한 train/test set에 대해 서로 다른 알고리즘으로부터 생성된 예측값을 이야기할 수 있다.
- 각 샘플 내에서는 독립적이어야 하며, 두 샘플은 동일한 모집단에서 추출되어야 한다.
- 두 샘플은 독립적이지 않기 때문에 Mann-Whitney U test로 검정할 수 없다.
- Mann-Whitney U test에서와 동일하게, 각 샘플은 20개 이상의 관측치를 포함해야 한다.
The Wilcoxon signed ranks test is a nonparametric statistical procedure for comparing two samples that are paired, or related. The parametric equivalent to the Wilcoxon signed ranks test goes by names such as the Student’s t-test, t-test for matched pairs, t-test for paired samples, or t-test for dependent samples.
— Pages 38-39, Nonparametric Statistics for Non-Statisticians: A Step-by-Step Approach, 2009.
from numpy.random import seed from numpy.random import randn from scipy.stats import wilcoxon # seed the random number generator seed(1) # generate two independent samples data1 = 5 * randn(100) + 50 data2 = 5 * randn(100) + 51 # compare samples stat, p = wilcoxon(data1, data2) print('Statistics=%.3f, p=%.3f' % (stat, p)) # interpret alpha = 0.05 if p > alpha: print('Same distribution (fail to reject H0)') else: print('Different distribution (reject H0)') - 두 샘플이 다른 어떠한 방식으로 인해 paired하게 측정된 데이터일 경우 사용한다.
- Friedman Test
- 둘 이상의 데이터 샘플에 대해 동일한 분포를 가지고 있는지를 검정할 수 있다.
- 샘플 간 paired한 관계가 있을 경우 사용할 수 있다. (e.g. repeated measures)
- 아래 설명할 Kruskal-Wallies H test의 generalized 버전이다.
The Friedman test is a nonparametric statistical procedure for comparing more than two samples that are related. The parametric equivalent to this test is the repeated measures analysis of variance (ANOVA). When the Friedman test leads to significant results, at least one of the samples is different from the other samples.
— Pages 79-80, Nonparametric Statistics for Non-Statisticians: A Step-by-Step Approach, 2009.
from numpy.random import seed from numpy.random import randn from scipy.stats import friedmanchisquare # seed the random number generator seed(1) # generate three independent samples data1 = 5 * randn(100) + 50 data2 = 5 * randn(100) + 50 data3 = 5 * randn(100) + 52 # compare samples stat, p = friedmanchisquare(data1, data2, data3) print('Statistics=%.3f, p=%.3f' % (stat, p)) # interpret alpha = 0.05 if p > alpha: print('Same distributions (fail to reject H0)') else: print('Different distributions (reject H0)') - Kruskal-Wallis H Test
- 둘 이상의 데이터 샘플에 대해 동일한 분포를 가지고 있는지를 검정할 수 있다.
- Mann-Whitney U Test의 generalized 버전이라고 볼 수 있다.
- 샘플들 간 paired 관계가 있을 경우 사용할 수 없다. (Friedman test로 검정한다.)
- 어떤 데이터 샘플 간, 얼마나 차이가 나는지는 확인할 수 없다.
When the Kruskal-Wallis H-test leads to significant results, then at least one of the samples is different from the other samples. However, the test does not identify where the difference(s) occur. Moreover, it does not identify how many differences occur. To identify the particular differences between sample pairs, a researcher might use sample contrasts, or post hoc tests, to analyze the specific sample pairs for significant difference(s). The Mann-Whitney U-test is a useful method for performing sample contrasts between individual sample sets.
— Page 100, Nonparametric Statistics for Non-Statisticians: A Step-by-Step Approach, 2009.
- 각 데이터 샘플은 5 이상의 관측치를 가져야 하며, 관측치들은 독립적이어야 한다. 데이터 샘플들의 관측치 수는 상이할 수 있다.
# generate three independent samples data1 = 5 * randn(100) + 50 data2 = 5 * randn(100) + 50 data3 = 5 * randn(100) + 52 from numpy.random import seed from numpy.random import randn from scipy.stats import kruskal # seed the random number generator seed(1) # generate three independent samples data1 = 5 * randn(100) + 50 data2 = 5 * randn(100) + 50 data3 = 5 * randn(100) + 52 # compare samples stat, p = kruskal(data1, data2, data3) print('Statistics=%.3f, p=%.3f' % (stat, p)) # interpret alpha = 0.05 if p > alpha: print('Same distributions (fail to reject H0)') else: print('Different distributions (reject H0)') - 둘 이상의 데이터 샘플에 대해 동일한 분포를 가지고 있는지를 검정할 수 있다.
- Multivariate Kruskal-Wallis test(MKW)
- He F, Mazumdar S, Tang G, Bhatia T, Anderson SJ, Dew MA, Krafty R, Nimgaonkar V, Deshpande S, Hall M, Reynolds CF 3rd. NONPARAMETRIC MANOVA APPROACHES FOR NON-NORMAL MULTIVARIATE OUTCOMES WITH MISSING VALUES. Commun Stat Theory Methods. 2017;46(14):7188-7200.
- 논문에서 Multivariate Kruskal-Wallis test와 MANOVA 검정을 비교하였다.
- MKW 검정에서는 MANOVA과 다르게 종속변수가 순서형일 때도 적용이 가능하다.
- missing data가 있는 경우 MCAR를 가정하여 결측치를 채운 후 수행할 수 있다.
- 아래의 코드(R)는 논문에서 제공한 코드이다.
library(lattice) library(Matrix) ################################ ###### 1. mult-KW funtion ###### ################################ multkw<- function(group,y,simplify=FALSE){ ### sort data by group ### o<-order(group) group<-group[o] y<-as.matrix(y[o,]) n<-length(group) p<-dim(y)[2] if (dim(y)[1] != n) return("number of oberservations not equal to length of group") groupls<-unique(group) g<-length(groupls) #number of groups# groupind<-sapply(groupls,"==",group) #group indicator# ni<-colSums(groupind) #num of subj of each group# r<-apply(y,2,rank) #corresponding rank variable# ### calculation of statistic ### r.ik<-t(groupind)%*%r*(1/ni) #gxp, mean rank of kth variate in ith group# m<- (n+1)/2 #expected value of rik# u.ik<-t(r.ik-m) U<-as.vector(u.ik) V<-1/(n-1)*t(r-m)%*%(r-m) #pooled within-group cov matrix Vstar<-bdiag(lapply(1/ni,"*",V)) W2<-as.numeric(t(U)%*%solve(Vstar)%*%U) ### return stat and p-value ### returnlist<-list(statistic=W2,d.f.=p*(g-1), p.value=pchisq(W2,p*(g-1),lower.tail=F)) if (simplify==TRUE) return (W2) else return (returnlist) } ########################################## ######## 2 MKW with missing values ####### ########################################## mkw.m<-function(group,y,r,weight){ ### count missng patterns ### p<-dim(y)[2] r.order<-r y.order<-y g.order<-group for (i in 1:p){ oo<-order(r.order[,i]) y.order<-y.order[oo,] g.order<-g.order[oo] r.order<-r.order[oo,] } J<-nrow(unique(r.order,MARGIN=1)) #number of missing patterns D<-data.frame(r.order) n<-length(group) ones<-rep(1,n) mc<-aggregate(ones,by=as.list(D),FUN=sum) #counts of each missing pattern mi<-mc$x pi<-p-rowSums(mc[,1:p]) ### get W^2_j ### W2<-rep(0,J) W2.c<-0 i.st<-1 for (j in 1:J){ i.end<-i.st+mi[j]-1 gg<-g.order[i.st:i.end] yy<-y.order[i.st:i.end,] ii<-mc[j,1:p]==F if (sum(as.numeric(ii))>0){ yy1<-as.matrix(yy[,ii]) if (mi[j]>pi[j]) W2[j]<-multkw(gg,yy1,simplify=T) ##### if mi[j]>p needs to dig more } if (prod(as.numeric(ii))==1) W2.c<-W2[j] i.st<-i.end+1 } if (weight=="prop") tj<-mi/sum(mi) else tj<-1/J W2<-sum(tj*W2) nu<-(W2)^2/sum((tj*W2)^2/pi/(g-1)) return(list(W2.m=W2,nu=nu,W2.c=W2.c)) } ######################################## ###### 3. monte carlo permutation ###### ######################################## multkw.perm<-function(nmc,group,y,r,weight){ ### count missng patterns ### p<-dim(y)[2] r.order<-r y.order<-y g.order<-group for (i in 1:p){ oo<-order(r.order[,i]) y.order<-y.order[oo,] g.order<-g.order[oo] r.order<-r.order[oo,] } J<-nrow(unique(r.order,MARGIN=1)) #number of missing patterns D<-data.frame(r.order) n<-length(group) ones<-rep(1,n) mc<-aggregate(ones,by=as.list(D),FUN=sum) #counts of each missing pattern mi<-mc$x W2.m.perm<-rep(0,nmc) W2.c.perm<-rep(0,nmc) stats0<-mkw.m(group,y,r,weight) W2.m<-stats0$W2.m W2.c<-stats0$W2.c nu<-stats0$nu for (i in 1:nmc){ i.st<-1 group.perm<-rep(0,n) group.perm<-sample(group,size=n) stats<-mkw.m(group.perm,y,r,weight) W2.m.perm[i]<-stats$W2.m W2.c.perm[i]<-stats$W2.c } p.mkw.m.perm<-sum(W2.m<W2.m.perm)/nmc p.mkw.m.chi2<-pchisq(W2.m,nu,lower.tail=FALSE) p.mkw.c.perm<-sum(W2.c<W2.c.perm)/nmc p.mkw.c.chi2<-pchisq(W2.c,p*(g-1),lower.tail=FALSE) return(list(W2.m=W2.m,p.mkw.m.perm=p.mkw.m.perm,p.mkw.m.chi2=p.mkw.m.chi2, p.mkw.c.perm=p.mkw.c.perm,p.mkw.c.chi2=p.mkw.c.chi2)) } ## 예시 group = data$예측분류 y = data %>% select(pqi, score, ipc_cnt) result = multkw(group,y,simplify=FALSE) print(result) ## 결과 # $statistic # [1] 1481.013 # # $d.f. # [1] 15 # # $p.value # [1] 6.106133e-307 - 참고

댓글남기기